02.Annotation
01.Introduction
我们十分关心找到的候选变异位于哪些基因上,对于个别变异来说,直接去NCBI genome browser上面搜索一下位置就可以,但对于大量的变异(比如GWAS关联出的候选基因),就需要用软件进行批量注释。ANNOVAR是一款用于变异注释的软件,使用简单方便,就以该软件为例讲讲怎么做变异注释,当然同类的软件还有SNPeff等,使用起来也都大同小异。
02.Download
ANNOVAR对个人及学术使用是免费的,只需要在他们网站上填写信息即可,注意邮箱最好是edu邮箱,不要填com后缀的。
提交后会收到类似下面这样的下载地址,直接下载即可。
http://www.openbioinformatics.org/annovar/download/0wgxR2rIVP/annovar.latest.tar.gz
此外,建库过程中还需要GtfToGenePred软件,直接官网下载。
03. Build annotation database
gtfToGenePred /home/guoyingwei/project/Reference/GCF_002263795.1_ARS-UCD1.2_genomic.rename.gtf -genePredExt UCD1.2_refGene.txt
# -geneNameAsName2 if specified, use gene_name for the name2 field instead of gene_id.
# 好像用直接下载好的GTF没问题,但如果使用GFF转换的GTF的话,gene id会显示数字,需要加这个参数
perl /home/guoyingwei/software/annovar/retrieve_seq_from_fasta.pl --format refGene --seqfile /home/guoyingwei/project/Reference/GCF_002263795.1_ARS-UCD1.2_genomic.addY.rename.fna UCD1.2_refGene.txt --out UCD1.2_refGeneMrna.fa
04.Annotation
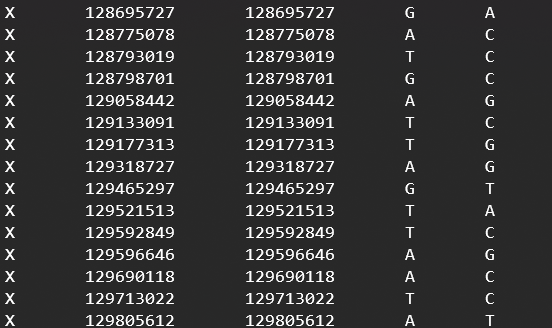
input文件5列,最后两列可以都是0。
perl /home/guoyingwei/software/annovar/table_annovar.pl -buildver UCD2.0 -protocol refGene -operation g -vcfinput TNFSF4.vcf.gz /home/guoyingwei/project/Reference/UCD2.0/annovar_db_UCD2.0/ -outfile TNFSF4.anno
perl /home/guoyingwei/software/annovar/table_annovar.pl -buildver UMD3.1 -protocol refGene -operation g sites_input /home/guoyingwei/project/Reference/annovar_db_UMD3.1/ -outfile sites_input